【6.7.3】GUIDE-Seq
GUIDE-Seq通过CRISPR-Cas核酸酶实现脱靶切割的全基因组分析
CRISPR RNA引导的核酸酶(RGN,RNA-guided nucleases)是广泛使用的基因组编辑试剂,但缺乏描述其全基因组脱靶切割活性的方法。在这里,我们描述了一种整体检测由RGN和潜在的其他核酸酶引入的DNA双链断裂(DSB,double-stranded breaks )的方法。这种方法称为全基因组无偏序测序(GUIDE-Seq),可通过捕获双链寡聚脱氧核苷酸而获得断裂。GUIDE-Seq在两个人类细胞系中的13个RGN上的应用表明,RGN的变异性很大目标活动和脱靶序列的未知特征。现有的计算方法或ChIP-Seq未检测到大多数已鉴定的位点。 GUIDE-Seq还确定了独立于RGN的基因组断点“热点”。最后,GUIDE-Seq显示,截短的引导RNA表现出显着减少的RGN诱导的脱靶DSB。我们的实验为迄今为止对RGN脱靶效应的全基因组鉴定定义了最严格的框架,并提供了一种在临床使用前评估这些核酸酶安全性的方法。
一、前言
CRISPR-Cas RGN是可靠的基因组编辑试剂,具有广泛的研究和潜在的临床应用。但是,RGNs在人类中的治疗性应用将需要全面了解其脱靶效应,以最大程度地降低有害结果的风险。化脓性链球菌Cas9核酸酶对DNA的切割是由可编程的〜100 nt向导RNA(gRNA)指导的。靶向是由gRNA 5’端的17-20 nts介导的,它们与“ protospacer” DNA位点互补紧邻5'-NGG形式的原间隔子相邻基序(PAM)。通过非同源末端连接(NHEJ)修复原间隔物中平末端Cas9诱导的DNA双链断裂(DSB)可以诱导可变长度的插入/缺失突变(indels)。我们的研究小组和其他研究人员先前已经表明,意外的RGN诱导的插入缺失可能发生在脱靶位点,该位点在原间隔子之间的差异多达五个位置,或者具有替代的PAM序列。另外,染色体易位可能是由于靶标和靶标以外的RGN诱导的裂解事件的结合所致。对于临床应用,即使是低频变化的鉴定也至关重要,因为使用RGN的离体和体内治疗策略预计需要对非常大的细胞群体进行修饰。甚至在罕见的细胞克隆亚群中引起致癌转化(例如,抑癌基因的失活突变或致瘤性染色体易位的形成)的诱导也引起特别关注,因为这种改变可能导致不良的临床结果。
鉴定可能在基因组中任何地方发生的插入缺失或高序重排是一项不容易解决的挑战,并且尚未描述用于在活细胞中无偏倚,全基因组范围内鉴定RGN诱导的脱靶突变的敏感方法,全基因组重测序已被用于尝试鉴定已编辑的单细胞克隆中的RGN脱靶变异[14,15],但是对大量基因组进行测序的极高预计成本使得该方法在寻找细胞群中低频率事件中不切实际。我们和其他人已使用集中深度测序来鉴定潜在脱靶位点的插入缺失突变,这些突变是通过与靶位点的序列相似性或通过从部分简并的结合位点文库中进行体外选择而鉴定的。但是,这些方法是有偏见的,因为它们假定脱靶序列与靶上位点密切相关,结果可能错过基因组中潜在的脱靶位点。 ChIP-Seq也已被用于识别与催化死亡的Cas9(dCas9)复合的gRNA的脱靶结合位点,但是大多数已发表的研究表明,这些位点中的极少数(如果有的话)表示通过活性Cas9核酸酶。
在这里,我们描述了GUIDE-Seq的开发,它使我们能够为人类细胞中的13种不同RGN生成全局特异性图谱。这些概况表明,脱靶DSB的总数对于单个RGN而言差异很大,并建议有关化脓性链球菌或其他物种的RGN特异性的广泛结论应基于大量gRNA的表征。我们的发现还扩大了可能发生脱靶效应的序列的范围和性质,并证明dCas9的ChIP-Seq和两种广泛使用的计算方法不能识别GUIDE-Seq所发现的许多位点。我们的方法还确定了RGN独立的DNA断点热点,这些热点可以与RGN诱导的DSB一起参与更高阶的基因组改变,例如易位。最后,我们在直接比较中显示,截断gRNA的原间隔区互补区可大大改善其全基因组脱靶DSB谱,证明GUIDE-Seq可用于评估旨在提高RGN特异性的技术进步。此处概述的实验提供了迄今为止描述的最严格的策略,用于评估可考虑用于治疗用途的RGN的特异性。
二、结果
2.1 GUIDE-Seq方法概述
GUIDE-Seq由两个阶段组成(图1a):
- 在阶段I中,在活的人类细胞基因组中钝端(blunt-ended)的RGN诱导的DSB通过在这些断裂处整合一个钝的双链寡聚脱氧核苷酸(dsODN)进行标记。 与NHEJ一致的末端连接过程。
- 在第二阶段中,使用无偏扩增和下一代测序技术,可在核苷酸水平上精确定位基因组DNA中的dsODN整合位点。
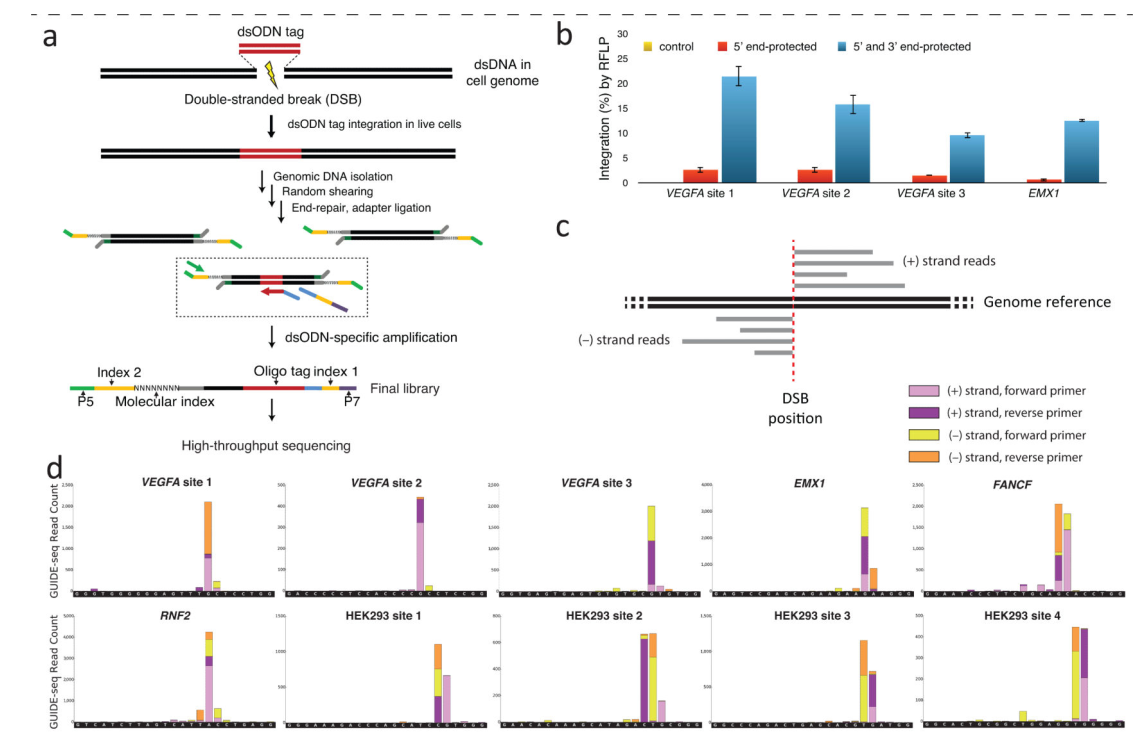



对于阶段I,我们优化了条件,以将钝的5’磷酸化的34 bp dsODN整合到人细胞中RGN诱导的DSB中。在最初的实验中,我们未能观察到将此类dsODN整合到RGN诱导的DSB中(数据未显示)。使用在两条DNA链的5’末端带有两个硫代磷酸酯键的dsODNs(旨在稳定细胞中的寡核苷酸),我们仅观察到适度的可检测整合频率(图1b)。但是,在两条链的5’和3’末端添加硫代磷酸酯键(在线方法)会导致强大的整合效率(图1b)。这些整合率仅比在这些位点(即在没有dsODN的情况下)仅由RGNs诱导的插入缺失的频率低2-3倍(数据未显示)。
对于第二阶段,我们制定了一种策略,使我们能够以无偏见的方式选择性地扩增和测序仅带有整合dsODN的片段(图1a)。我们首先通过将“单尾”下一代测序适配器连接到用dsODN转染的细胞和编码RGN组分的质粒中的随机剪切的基因组DNA来实现此目的。然后,我们进行了一系列的PCR反应,这些反应由一个特异于dsODN退火的引物和另一个与测序衔接子退火的引物启动(图1a和补充图1)。由于测序适配器仅是单尾的,因此可以对dsODN附近的序列进行特定的单向扩增,而没有诸如线性扩增介导的(LAM)-PCR21、22等方法固有的偏倚和背景。作为单尾适配器/标签(STAT)-PCR方法。通过使用与dsODN中每条链退火的引物进行STAT-PCR反应,我们获得了每个整合标签两侧的相邻基因组序列的读数(图1c)。在扩增过程中加入随机的8 bp分子条形码(补充图1)可以校正PCR偏差,从而可以准确定量从高通量测序获得的独特测序读数(补充方法)。
2.2 人类细胞中RGN的全基因组脱靶裂解谱
我们用Cas9和针对U2OS或HEK293人类细胞系中各种内源人类基因的十种不同gRNA进行了GUIDE-Seq(补充表1)。通过分析dsODN整合位点(补充方法),我们能够确定由10个RGN的每一个诱导的DSB的精确基因组位置,并映射到核苷酸水平(图1d和补充图2)。对于> 80%的这些基因组位点,我们能够鉴定出重叠的靶序列,即靶上位点或密切相关的靶外位点(补充方法)。我们为每个RGN识别出的脱靶位点的总数变化很大,范围从零到> 150(图1e),这表明在任何极端情况下,任何特定RGN进行的有害基因组切割都可能是相当大的或很小的。对照实验中,我们使用基于锚定多重PCR(AMP)的下一代测序技术对了五个RGN的靶位和离靶位点处的dsODN插入进行了测序(图3a,在线方法和补充方法),结果显示> 93这些位点中的百分比(132个中的123个)显示出可检测到的dsODN分子之一,这与NHEJ介导的DSB捕获相符(数据未显示)。
我们没有观察到通过GUIDE-Seq观察到的脱靶位点总数与脱靶位点相对于人类基因组的正交性或脱靶前靶标序列的GC含量之间存在任何明显的相关性(图1f和图1f)。 1克)。 发现脱靶序列分散在外显子,内含子和非编码基因间区域(图1i)的整个基因组中(图1h和补充图3)。 我们确定的脱靶序列中包括先前已知的四个RGN4、5(图1e,2a-2j和补充表2)中所有28个真正的脱靶位点。 GUIDE-Seq还确定了许多先前未知的脱靶位点,这些位点位于整个人类基因组中(图1e,1h,1i,2a-2j,补充表2和补充图3)。


接下来,我们测试了由GUIDE-Seq识别的每个脱靶位点的测序读取数(如图2a-2j所示,以下称为“ GUIDE-Seq读取计数”)是否代表插入缺失的相对频率仅由RGN(即在不存在dsODN的情况下)会诱发的信号。我们使用AMP测序检查了仅表达核酸酶成分的细胞中的这些相同位点,发现> 80%(132个中的106个)具有NHEJ介导的RGN裂解事件修复的可变长度插入缺失。支持我们的结论,即GUIDE-Seq可以识别出真正的RGN脱靶位点(图3a和补充图4)。 (许多我们未见插入缺失证据的位点的GUIDE-Seq读取计数也很低,这表明无法在这些位点检测突变可能与测序的敏感性和实验的采样深度有关)。我们检测到的插入缺失突变频率范围为0.03%至60.1%。值得注意的是,我们观察到所有五个RGN脱靶位点的GUIDE-Seq读数计数与indel突变频率之间呈线性正相关(图3b-3f)。因此,我们得出结论,给定位点的GUIDE-Seq读计数代表了RGN对该序列的切割效率的定量测量。


2.3 RGN诱导的脱靶序列特征分析
通过目视检查,我们通过GUIDE-Seq对9个RGN鉴定了脱靶位点,突显了这些核酸酶可裂解的变异序列的多样性。这些位点在原间隔区序列中包含多达6个错配(与先前的报告显示,体外裂解携带多达7个错配的位点一致),非规范的PAM(包括先前描述的NAG和NGA序列5、23,还有NAA,NGT ,NGC和NCG序列),以及在gRNA / protospacer界面处出现1 bp的“凸出”型错配(图2a-2j)。原间隔物不匹配往往发生在目标位点的5’末端,但也可能在某些3’末端位置发现,这支持以下概念:没有简单的规则可基于position预测不匹配效应。值得注意的是,某些脱靶位点实际上比其匹配的脱靶位点具有更高的测序读取计数(图2a-2c,2i),这与我们之前的观察结果一致,即脱靶突变频率在某些情况下可能高于预期的目标站点脱靶突变频率。。四个RGN的许多先前已知的脱靶位点都是具有高读取计数的位点(图2a-d),这表明较早的分析主要鉴定出了最有效切割的位点。
对9个RGN的GUIDE-Seq数据进行的定量分析使我们能够量化不同变量(例如错配数,位置和类型)对脱靶位点切割的潜在贡献和影响。我们发现,带有一定数目的原间隔子错配的总基因组位点中,被RGN切割的比例随着错配数量的增加而降低(图3g)。另外,序列读取计数显示总体下降趋势,错配数量增加(图3h)。通常,与靶标5’端较近的原间隔区错配比与3’端较近的GUIDE-Seq读计数下降幅度较小,尽管距PAM 1至4 bp的错配有些比5到8 bps的耐受性更好(图3i)。不匹配的性质还与对GUIDE-Seq读取计数的影响有关。摆动不匹配经常发生在脱靶位点,我们的分析表明,与其他非摆动不匹配相比,摆动不匹配对GUIDE-Seq读数计数的影响较小(图3j)。与这些结果一致,我们发现在单变量回归分析中解释脱靶裂解最大变异程度的单个因素是错配数,位置和类型。相比之下,其他因素(例如近端PAM序列的密度,基因表达水平或基因组位置(基因间/内含子/外显子))解释了GUIDE-Seq裂解阅读计数变异的比例小得多(图3k)。一个综合的线性回归模型考虑了多个因素,包括错配位置,错配类型,基因表达水平和近端PAM序列的密度,得出的结果与单变量分析一致(补充图5)。这项分析还使我们能够独立地估计,平均而言,根据其位置,每个额外的摆动不匹配都会使脱靶切割率降低大约2-3倍, 而其他的非摆动失配会使切割速率降低大约三倍(补充图5)。
2.4 与计算机模拟脱靶预测方法的比较
建立了GUIDE-Seq的功效后,我们接下来将我们的方法与两种流行的预测脱靶突变位点的计算程序进行了直接比较:MIT CRISPR设计工具( http://crispr.mit.edu )和E-CRISP软件( http://www.e-crisp.org/E-CRISP/ )。这两个程序都基于有关错配数量和位置的“规则”来识别潜在的脱靶站点。在直接比较中,我们发现这两个程序都未能识别出GUIDE-Seq发现的针对9个RGN的绝大多数脱靶站点(图4a和4b)。由于E-CRISP和MIT程序根本没有考虑分别具有3个和4个以上不匹配的脱靶,因此错过了许多此类站点(图4c和4d)。即使在所考虑的序列中,这些程序仍未能识别出大多数真正的脱靶位点(图4c和4d),突显了它们目前对决定是否发生切割的因素的能力有限。特别值得一提的是,尽管MIT计划分配的排名得分确实在其正确识别的站点子集中具有一定的预测能力,但错过的站点包括只有一个不匹配的站点(图4c和4d)。
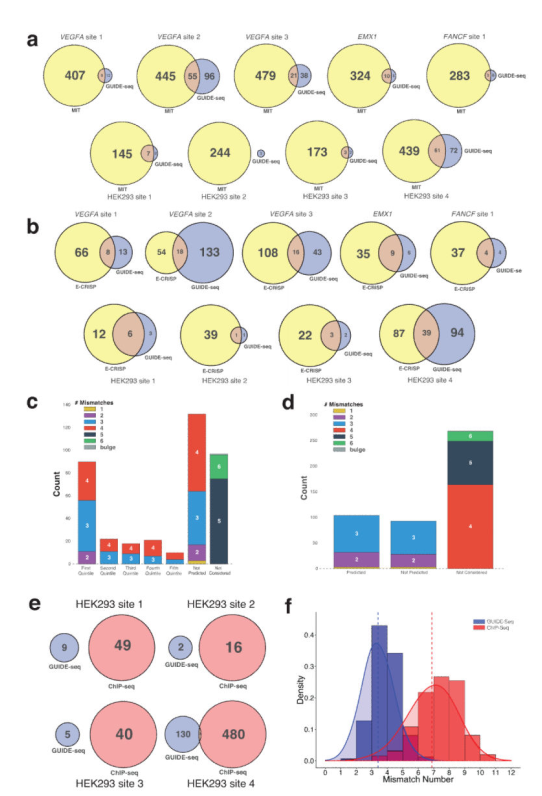

2.5 与ChIP-Seq发现的脱靶结合位点比较
我们还试图将GUIDE-Seq与先前描述的用于识别Cas9结合位点的ChIP-Seq方法进行比较。我们通过GUIDE-Seq评估的四个RGN使用了先前在ChIP-Seq实验中具有催化活性的Cas9(dCas9)表征的gRNA。 GUIDE-Seq鉴定的Cas9脱靶切割位点与ChIP-Seq鉴定的dCas9脱靶结合位点之间几乎没有重叠。在我们为这四个gRNA鉴定的149个RGN诱导的脱靶切割位点中,只有三个通过先前使用相同gRNA的dCas9 ChIP-Seq实验被鉴定出来(图4e)。缺少重叠可能是因为dCas9脱靶结合位点与Cas9脱靶裂解位点根本不同,这一假设得到了我们的数据支持,表明GUIDE-Seq鉴定出的这四个gRNA的Cas9脱靶裂解位点平均具有与ChIP-Seq鉴定的结合位点相比,错配要少得多(图4f),而先前的研究结果表明,很少有dCas9结合位点在存在活性Cas916-19的情况下显示出插入缺失的证据。尽管GUIDE-Seq无法识别先前由ChIP-Seq识别并报告为Cas9诱变的靶标的七个脱靶位点,但我们认为这是因为这些位点在该较早的研究中被误认为是真正的脱靶裂解。位点(补充结果和补充图6)我们得出结论,通过ChIP-Seq发现的极少(如果有)dCas9脱靶结合位点实际上代表了真正的Cas9脱靶裂解位点。
2.6 由GUIDE-Seq识别的与RGN无关的DSB热点
我们的GUIDE-Seq实验还揭示了用于我们研究的U2OS和HEK293细胞中总共存在30个独立于RGN的DSB热点(补充表3)。 在分析来自U2OS和HEK293细胞对照实验的基因组DNA时,我们发现了这些基因,在这些实验中,我们仅转染了dsODN而没有RGN编码质粒。 与RGN诱导的DSB精确映射到特定碱基对位置不同,RGN无关的DSB热点具有dsODN整合模式,该模式在出现它们的每个位点上分布更广泛(补充方法)。 这30个断点热点分布在许多染色体上,似乎存在于着丝粒或端粒区域或附近(图5a)。 这些热点中只有两个是两种细胞系共有的,而大多数似乎是细胞系特异性的(U2OS中为25个,HEK293细胞中为七个)(图5a和补充表3)。
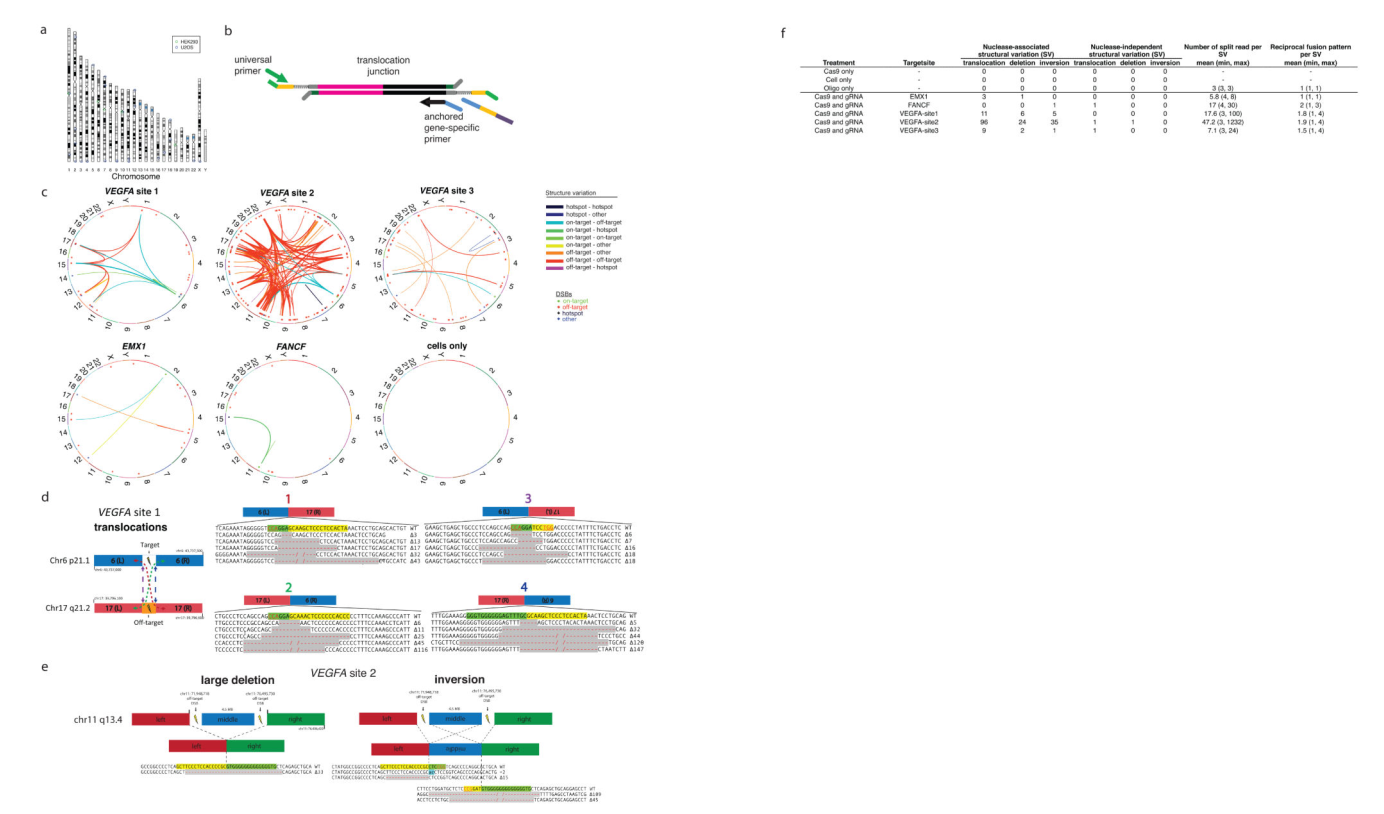

2.7 大规模基因组重排分析
在分析旨在识别RGN诱导的和RGN独立的DSB插入缺失的基于AMP的测序实验结果的过程中,我们还发现这些断裂中至少有一些可以参与易位,倒位和大缺失。 AMP方法使我们能够观察到这些大规模的基因组改变,因为,对于每个检查的DSB位点,它使用的巢状基因座特异性引物仅锚定在一个固定端,而不是一对侧翼基因座特异性引物(图5b)。
对于我们检查的五个RGN,AMP测序显示RGN诱导的靶上和靶外DSB可能参与多种易位(图5c)。在至少一个案例中,我们观察到了由一对DSB引起的所有四个可能的易位事件(图5d)。当在同一条染色体上存在两个DSB时,我们还观察到了大的缺失和倒位(图5c)。我们还观察到两个RGN诱导的断裂之间大量缺失以及相同插入序列倒置的例子(图5e)。值得注意的是,我们的结果还揭示了RGN诱导的和RGN独立的DSB之间的易位(以及缺失或倒位)(图5c和5f),这表明在评估脱靶效应时需要考虑这两种类型的断裂之间的相互作用。细胞基因组上的RGNs。尽管我们的数据表明,这些大规模基因组重排的频率可能很低,但是利用我们现有数据集的测序深度不可能进行精确的定量。增加测序读数的数量应增加检测的灵敏度,并使这些重要的基因组改变得到更好的定量。
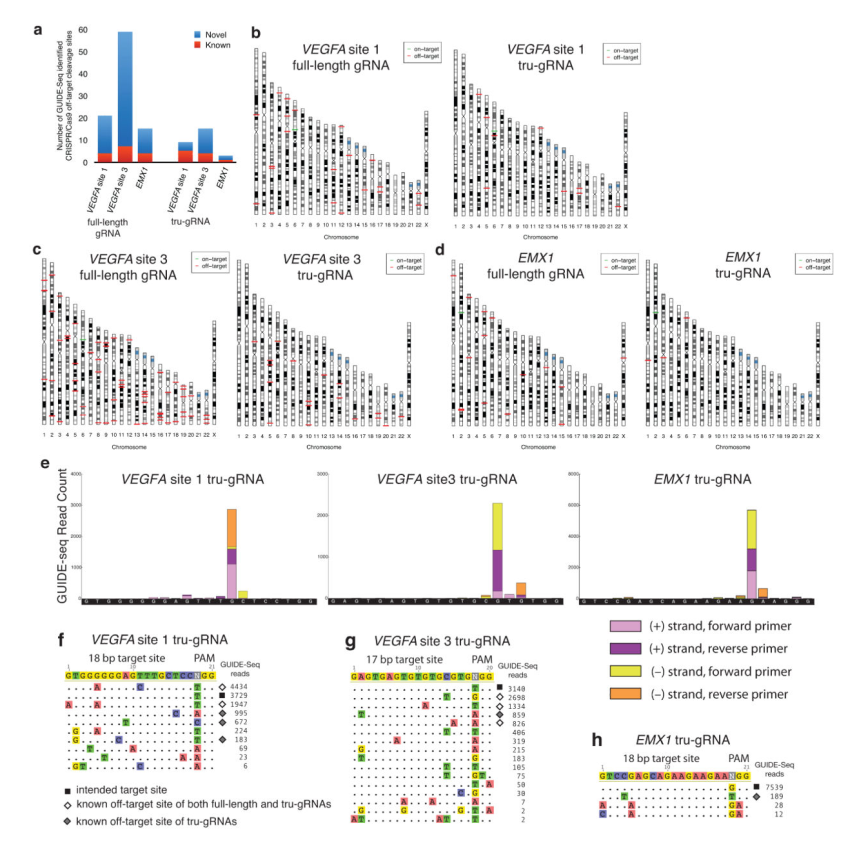
2.8 截短的gRNA指导的RGN的GUIDE-Seq谱
我们小组先前的研究表明,使用带有17或18个核苷酸的截短互补区的gRNA可以减少全长gRNA定向的RGN已知脱靶位点的突变频率。但是,由于该分析仅限于少数已知的脱靶位点,因此在我们较早的实验中,这些截短的gRNA(tru-gRNA)的全基因组特异性仍然不确定。我们使用GUIDE-Seq获得了由三个tru-gRNA指导的RGN的全基因组DSB谱,每个trug是我们上面分析的三个全长gRNA的较短版本。在所有三种情况下,使用tru-gRNA时,由GUIDE-Seq识别的脱靶位点的总数均显着降低(图6a-6d)。 GUIDE-Seq读图的映射使我们能够精确识别目标上(图6e)和目标外(补充图7)位点的切割位置。正如预期的那样,脱靶位点列表中包括三个tru-gRNA定向的12个先前已知的RGN脱靶位点中的10个(图6f-6h)。我们确定的脱靶位点序列在原间隔物中主要具有一或两个错配,但有些位点却多达四个(图6f-6h)。另外,某些位点具有NAG,NGA和NTG形式的替代PAM序列(图6f-6h)。这些数据在全基因组范围内提供了证实,即gRNA的截短可以显着降低RGN的脱靶效应,并显示GUIDE-Seq如何可用于评估RGN平台的特异性改善。

三、讨论
我们的研究表明,GUIDE-Seq为检测RGN诱导的DSB提供了一种无偏见,全基因组的灵敏方法。该方法是无偏见的,因为它在检测DSB时无需假设脱靶位点的性质(例如,假设脱靶位点与脱靶位点的序列密切相关)。 GUIDE-Seq在全基因组范围内(包括外显子,内含子和基因间区域内)识别脱靶位点。尽管当前缺乏用于全面识别细胞中所有RGN脱靶位点的金标准方法使我们无法确定性地了解GUIDE-Seq的敏感性,但我们认为,对于以下情况,其假阴性率很低原因:
- 首先,所有RGN诱导的平末端DSB均应通过NHEJ摄取平末端dsODN,这一假设得到了我们观察到的GUIDE-Seq读数计数(用于测量dsODN摄取)和插入缺失频率之间强相关性的支持RGN(可测量DSB形成的速率及其诱变修复)(图3b-3f)。我们注意到这些相关性包括超过130个位点,这些位点显示了广泛的indel诱变频率。
- 其次,使用先前确定的脱靶位点作为基准(这是目前评估成功的唯一方法),GUIDE-Seq能够检测出其中40个位点中的38个,这些位点的诱变频率范围可低至0.12%。该方法检测了四个完整的gRNA的全部28个先前已知的脱靶位点,以及三个tru-gRNA的12个先前的已知脱靶位点中的10个(有关为什么我们没有检测到40个中的两个的潜在解释,请参见补充讨论)网站)。
尽管我们的验证实验表明GUIDE-Seq可以灵敏地检测被RGN诱变的脱靶位点,其频率低至0.1%,但其检测能力可能会随着更深的测序而进一步提高。使用下一代测序技术检测插入缺失的策略受到平台错误率(通常为〜0.1%)的限制。相比之下,GUIDE-Seq使用测序来识别dsODN插入位点而不是插入缺失,因此不受错误率的限制,但受测序深度的限制。例如,我们相信在我们的GUIDE-Seq实验中检测到的少数位点在序列验证实验中未发现插入缺失,实际上代表的可能是插入缺失突变频率低于0.1%的位点。与此相符,我们注意到这26个位点中除三个位点之外的所有位点的GUIDE-Seq读计数均低于100。这些观察结果表明,我们可以简单地通过增加测序读数来提高GUIDE-Seq的灵敏度。 (并通过增加用作扩增模板的基因组数量)。例如,使用产生1000倍以上读数的测序平台,将能够检测出诱变频率降低三个数量级(即0.0001%)的位点,并且随着下一代测序的不断改进,我们预计会进一步增加技术。值得注意的是,即使我们第二次重复了GUIDE-Seq实验(数据未显示),我们评估的RGN之一也没有产生任何可检测到的脱靶效应。这一发现提出了一种有趣的可能性,即某些gRNA可能诱导很少的突变,或者可能不会诱导不期望的突变(至少在这些GUIDE-Seq实验的当前检测极限下)。
在直接比较中,我们发现两个现有的计算程序无法识别GUIDE-Seq找到的大多数真正的脱靶位点。考虑到这些程序所使用的参数是基于对脱靶位点的性质的限制性更强的假设而得出的,这些假设不能解释更多的原间隔物不匹配(最多六个)以及我们的GUIDE识别出的新的替代PAM序列,这并不完全令人惊讶-Seq实验。将来可能会开发出更好的预测程序,但这样做将需要实验确定的基因组范围的脱靶位点,以用于更多的RGN。在开发此类程序之前,将通过GUIDE-Seq等实验方法最有效地解决目标外站点的识别问题。
我们的实验结果阐明了dCas9的脱靶结合位点与Cas9的脱靶裂解位点之间的明显区别。我们的结果强烈表明,用ChIP-Seq捕获的dCas9与脱靶位点的结合代表了与Cas9核酸酶裂解脱靶位点不同的生物学过程,这与最近的研究结果表明为了有效切割,需要使用带有原间隔子的gRNA的'5-端。尽管ChIP-Seq分析无疑将在表征dCas9融合蛋白的全基因组结合中发挥作用,但该方法显然无法有效地确定催化活性RGN的全基因组脱靶切割位点。
GUIDE-Seq与其他先前描述的全基因组识别细胞中DSB位点的方法相比,具有许多优势。 BLESS(断裂标记,链霉亲和素富集和下一代测序)寡核苷酸标记方法是在固定的,可透化的细胞上原位进行的。除了容易出现与细胞固定相关的伪影外,BLESS仅捕获单个时刻存在的中断。相比之下,GUIDE-Seq在活细胞上执行,并捕获在更长的时间段(天)内发生的DSB,从而使其更加灵敏和全面。将整合缺陷型慢病毒(IDLV)DNA捕获到DSB附近的区域中并通过LAM-PCR鉴定这些基因座已用于鉴定工程化锌指核酸酶(ZFN)和转录激活因子样的少量脱靶位点人细胞中的效应子核酸酶(TALENs)。但是,IDLV整合事件的数量通常很少,并且分散在距离实际脱靶DSB的500 bps的距离内,这使得精确绘制裂解事件的实际的脱靶站点位置并推断其序列具有挑战性。。此外,先前IDLV捕获实验中使用的LAM-PCR过程还存在序列偏倚和/或有用测序读取效率低的问题。总的来说,这些局限性也可能解释了以前的研究中显然无法检测较低频率的ZFN脱靶位点。相比之下,dsODNs通过GUIDE-Seq非常高效且精确地集成到了DSB中,从而能够以单核苷酸分辨率绘制断裂图谱,并能够简单,直接地鉴定核酸酶脱靶切割位点。此外,与LAM-PCR相比,我们的STAT-PCR方法可对dsODN已整合的基因组DNA片段进行高效,无偏扩增和测序。我们注意到,STAT-PCR可能会超出其在GUIDE-Seq中的用途(例如,在全基因组范围内绘制病毒整合位点)。
GUIDE-Seq还确定了即使在没有RGN的情况下,细胞中也会出现的断点热点。我们认为这些DSB不仅是GUIDE-Seq的产物,因为我们基于AMP的测序实验不仅验证了dsODN的捕获,而且验证了indel的形成(数据未显示)以及涉及这些位点的大规模基因组重排。值得注意的是,在我们的研究中,我们发现的大多数热点似乎对两种细胞系都是唯一的,但两种似乎都相同。在将来的研究中,定义控制为什么某些站点是一种细胞类型而不是另一种细胞类型的断点热点的参数将是有趣的。同样,因为我们的结果表明这些断点热点可以参与易位,所以特定于细胞类型的断点热点的存在可能有助于解释为什么某些基因组重排仅在特定的细胞类型中发生而在其他细胞类型中不发生。据我们所知,GUIDE-Seq是第一种被描述的方法,它可以识别活人细胞中的断点热点,而无需添加抑制DNA复制的药物(例如,蚜虫碱)。因此,我们希望它将为识别和研究这些中断提供有用的工具。
我们的工作为鉴定RGN引起的易位建立了重要的定性方法。 GUIDE-Seq发现的基于AMP的RGN诱导和RGN独立DSB的靶向测序可以发现涉及这两种位点的大规模基因组重排(易位,缺失和倒位),突出了检查所有这些基因座的重要性。此外,大概不是所有RGN诱导的或RGN独立的DSB都会参与大规模变更,而了解为什么某些站点会这样做而其他站点却不会导致这些重排将是进一步研究的重要领域。
GUIDE-Seq也将提供一种重要的方法来评估全基因组范围内RGN平台的改变。在本报告中,我们使用了GUIDE-Seq来显示使用截短的gRNA可以减少全基因组的脱靶效应。我们设想,GUIDE-Seq也可用于评估来自其他细菌或古细菌的替代Cas9核酸酶的特异性。 GUIDE-Seq也可能适用于评估核酸酶的全基因组特异性,例如二聚ZFN,TALEN和CRISPR RNA引导的FokI核酸酶(RFN),它们产生5’突出端或成对的Cas9切口酶,它们产生5’突出端或3-overhangs。在初步实验中,我们已经表明,blunt dsODN可以捕获到ZFN,TALEN和RFN诱导的断裂中(数据未显示)。但是,扩展GUIDE-Seq以检测其他类型的DSB无疑将要求对dsODN进行其他修改,以优化其有效捕获此类中断的能力。
我们希望我们使用GUIDE-Seq和基于AMP的测序的整体方法将对评估RGN引起的脱靶突变和基因组重排非常有用。 GUIDE-Seq最有可能扩展为在NHEJ处于活动状态并且可以有效地引入所需组件的任何单元中使用;例如,我们已经在人K562和小鼠胚胎干细胞中实现了有效的dsODN整合(数据未显示),在未转化的原代细胞中进行该方法的未来实验将引起极大兴趣。此处概述的策略可以用作严格的临床前途径的一部分,以客观地评估提议用于治疗用途的任何RGN的潜在脱靶作用,从而从根本上改善最终将这些试剂翻译至临床的前景。
四、讨论
。。。
参考资料
- GUIDE-Seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases Shengdar Q. Tsai, Zongli Zheng, Nhu T. Nguyen, Matthew Liebers, Ved V. Topkar, Vishal Thapar, Nicolas Wyvekens, Cyd Khayter, A. John Iafrate, Long P. Le, Martin J. Aryee, J. Keith Joung .Nat Biotechnol. Author manuscript; available in PMC 2015 Aug 1. Published in final edited form as: Nat Biotechnol. 2015 Feb; 33(2): 187–197. Published online 2014 Dec 16. doi: 10.1038/nbt.3117
